ショウジョウバエの遺伝子制御、胚全体のモデル化に成功

生物のゲノム解読が急速に進む一方で、その遺伝情報を基に多細胞生物が受精卵からどのように発生してくのかはまだよくわかっていません。OIST ゲノム・遺伝子制御システム科学ユニット(ニコラス・ラスカム 教授)で研究するガース・イルズリー博士はハーバード大学医学大学院のAngela DePace助教とともにこの問題に挑みました。本研究では、統計的な手法を用いてショウジョウバエの初期の発生過程をモデル化し、胚全体の遺伝子発現を正確に予測することに成功しました。本研究はオープンアクセス誌eLIFEで8月6日に発表されました。
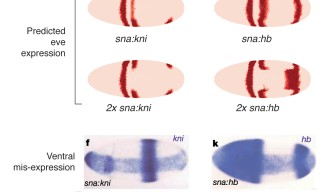
ショウジョウバエの体節(昆虫の前後に沿った繰り返し構造)を作るのに重要な遺伝子の1つにeven skipped (eve)という遺伝子があります。この遺伝子は発生の初期に7本の帯となって発現します。一般的に遺伝子の発現は転写因子と呼ばれるタンパク質により抑制されたり、促進されたりします。eve遺伝子が発現するときの胚は約6000個の細胞で構成されていますが、これまでの計算モデルはこのときの胚全体でのeve遺伝子の発現を正確にシミュレーションすることができず、その理由はまだ発見されていない重要な要素をモデルに組み込めていないからではないかと考えられていました。イルズリー博士は、実際に計測されたショウジョウバエの胚のデータ(Berkeley Drosophila Transcription Network Project)を使って、転写因子の濃度から発現を予測するより簡潔なモデルを構築し、胚全体の計算を初めて可能にしました。本研究では、eve遺伝子の2、3および7番目の帯での発現を制御するDNA上の2つの領域に着目して計算を行ったところ、eve遺伝子の発現を正確に予測することができました。また構築されたモデルを用いて、突然変異や異所性発現による転写因子の変化がeve遺伝子の発現に与える影響を帯の形状で正確に再現しました(図1)。
このモデルは、まだ解明されていない仮説の検証や、実際の実験結果との比較に用いることで、生物学上の新しい発見を促進するものと期待されます。イルズリー博士は、今後脊索動物のホヤなどの生物を使って、他の生物にも適用できるようにモデルを発展させたいと考えています。