生命の本を読む
数世紀前の生物学者の目には、生物の世界は混乱してしまうほど多様に映ったことでしょう。バクテリアや盆栽の木とネズミやキノコの間には、生きているという事実以外に何か共通点を見出すことができたでしょうか?しかし、19世紀から20世紀にかけて生物学的発見が相次ぎ、科学者たちは、この地球上のすべての細胞生物が共通して持っているものー共通の祖先から受け継がれた言語ーを発見しました。それは、英語や日本語のような言語ではなく、遺伝子の言語です。
DNA分子の中に書き込まれているこの遺伝子言語は、表面的には一見単純なように見えます。結局のところ、DNAはA、T、C、Gのたった4つの文字、つまり塩基からできているのです。この遺伝子の文字が長く連なってできた文章で構成される遺伝子は、命令系統として機能し、体内の細胞にRNAやタンパク質を送り出すように指令を出しています。そして、すべてのDNAが合わさってゲノムを形成し、各生物の発生や生存において中心的な役割を果たしているのです。
ゲノムの解読と生物間での遺伝子配列の比較は、生物学的・計算科学的分野における20世紀最大の課題の一つでした。その最前線で活躍する科学者の一人が、昨年、沖縄科学技術大学院大学(OIST)の教授(アジャンクト)に着任したジーン・マイヤーズ博士です。
「私はコンピュータサイエンティストですが、特に生物学的な課題を解決するために必要なコンピュータ技法に関心を向けています」とジーン・マイヤーズ教授は述べています。

バイオインフォマティクスの先駆け
マイヤーズ教授は、バイオインフォマティクス(生物情報科学)の先駆者の一人として世界でも著名な科学者です。バイオインフォマティクスとは、DNA配列のような大規模で複雑な生物学的データをコンピュータで処理する比較的新しい分野です。
マイヤーズ教授が最初にバイオインフォマティクスに触れたのは1970年代後半で、コロラド大学の博士課程学生の時で、「当時はまだ名前がついていないほど新しい分野でした」と教授は説明しています。しかし、彼のキャリアを決定づけた最初の功績は、アリゾナ大学で勤務していた1990年に、BLAST(Basic Local Alignment Search Tool)というDNA配列の比較や識別に使用するソフトウェアの核となるアルゴリズムを開発したことでした。
従来よりも高速で正確なアルゴリズムを持つBLASTを開発したことで、マイヤーズ教授は大きな偉業を達成しました。しかし、当の本人はそのことに気づいていなかったようで、「BLASTがこれほどまでに引用されていることを知ったのは、ずっと後になってからのことです」と教えてくれました。
89,000以上の引用数を誇るBLASTは、引用が史上最も多い10の科学論文の一つであり、世界中の科学者にとって不可欠な研究ツールとなっています。
BLASTでの実績により、バイオインフォマティクス分野での知名度が高まったマイヤーズ教授は、自身のキャリアの中で最も野心的でリスクの高いプロジェクトに着手しました。それは、「ペアエンドショットガンシーケンシング」という解析法を使ったヒトゲノム解析です。
当時、DNAの文字、つまり塩基の解読は、100~1000塩基の比較的短い断片でしか行えませんでした。一方、ヒトゲノムはその何百万倍もの大きさで、32億塩基にも及びます。
この問題に対してマイヤーズ教授が提唱した解決策の一つが、「ペアエンド」ショットガンシーケンシングです。これはショットガンシーケンシングを改良したもので、ヒトゲノムのようにDNA配列が反復する領域を持つゲノムを解読するのに適しています。
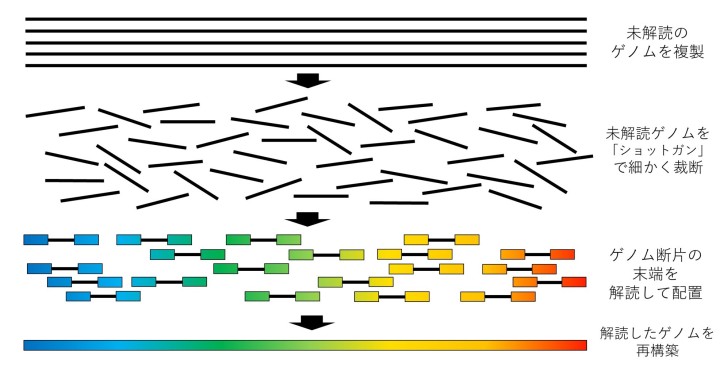
「ショットガンシーケンシングは小さなゲノムに使用されていましたが、ヒトゲノムほどの規模では特に反復DNA配列の量を考慮すると、対応できないと誰もが言っていました。私たちは、どのようにしてそれを行うかについてシミュレーションを使った論文を書いたのですが、1年半も出版できず、その後ようやく私たちに対する反論の論文と一緒に出版されました。誰もがこの研究を敬遠していましたが、私は何かを掴みかけていると思っていました」とマイヤーズ教授は振り返ります。
マイヤーズ教授がようやくチャンスに恵まれたのは、1998年に民間のバイオテクノロジー企業Celera Genomics社が設立されてからのことでした。Celera社の支援を得た教授は、ヒトゲノムのショットガンシーケンシングに賭け、大学を離れました。
「成功するという保証はありませんでしたが、これに賭けてみようと思いました。そして、その後どうなったかはご存じの通り。うまくいきました。」
ペアエンドショットガン解析により、ヒトゲノムの解読は予定よりも数年前倒しで行われ、費用は1億ドルで済みました。これは、公的資金を投入したヒトゲノム計画に要した30億ドルに対してほんのわずかな額です。
しかし、マイヤーズ教授は自身の功績について謙虚に語っています。「私はこのようなキャリアを歩むことになるとは夢にも思っていませんでした。とても恵まれていると思います。研究で大切なのは、準備を整えておき、ワクワクするようなチャンスがやってきた時に逃さないことだと思います。」
完璧なゲノムの構築
現在、マイヤーズ教授は学術界に戻り、ドイツのドレスデンにあるマックス・プランク分子細胞生物学・遺伝学研究所およびCenter for Systems Biology Dresdenの所長を務めています。そこでは、イメージングに焦点を当て、細胞や生物の中をより高い解像度で観察できる顕微鏡を開発しています。
しかし、ロングリードシーケンシングとして知られる最新世代のシーケンシング技術の登場により、教授はゲノム構築に再び惹かれており、OISTではそれが彼の研究の要となる予定です。
「新しいシーケンシング機器は、これまでよりもはるかに長いDNA断片をより高い精度で読み取ることができます」と教授は嬉しそうに語ります。これにより、遺伝子がどこに位置しているかという事前情報が無くても、ゼロから容易に新しいゲノムを再構築できるようになると説明しています。
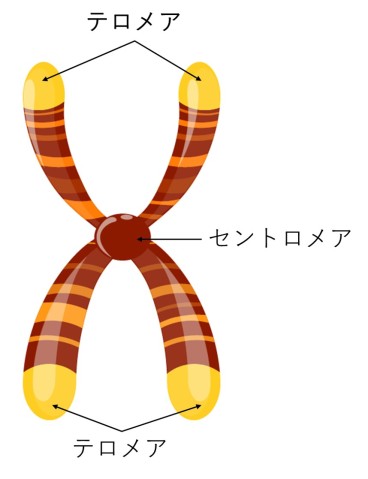
ロングリードシーケンシングが登場するまで、科学者たちは真に完全なゲノムを構築することができませんでした。ゲノムの一部の領域、例えば染色体の末端や中央にあるテロメアやセントロメアのような領域は、ショットガンシーケンシングで組み立てるのがあまりにも困難でした。
「私たちにはほとんどの遺伝子が分かっていましたが、その順序はよく分かっていませんでした。しかし、進化に関心があり、ゲノムがどのように変化するかを知りたければ、全ゲノムを知る必要があります。今、私たちは生物から血液や組織の検体を採取してゲノム配列を読み取り、この10年で構築されたヒトゲノムよりも質が高く、今後長く使い続けられるゲノムを再構築することができます」とマイヤーズ教授は述べています。
教授は、OISTで最新のシーケンシング技術を駆使してアルゴリズムを改良し、ゲノムの完璧な再構築に近づきたいと考えています。
マイヤーズ教授の新しい研究室である生態・進化ゲノミクス アルゴリズムユニットは、2名のポスドク研究員と、科学計算及びデータ解析セクションとDNAシーケンシングセクションに所属する技術者が2名在籍する小規模で的を絞ったグループになる予定です。2名の技術者は、OISTのあらゆる生物学分野の科学者にとって有益となるシーケンシングとバイオインフォマティクスのサービスを提供することになります。
マイヤーズ教授は、バイオインフォマティクスの学際的で協力的な性質を非常に重要なものと捉えています。「生物学者の科学研究を向上させるために使ってもらえるツールを生み出すことが私の務めだと考えています。そして、それを確実に実現する方法の一つが共同研究への参加です。」
教授は、その目的に向けてOIST内の3つのユニットと共同研究を行っています。その一つは、マリンゲノミックスユニットを率いる佐藤矩行教授との生態学的な研究です。同研究では、オニヒトデのゲノム配列の解析に取り組んでいます。オニヒトデはサンゴの捕食者で、爆発的に増殖してサンゴ礁を広範囲にわたって荒廃させるおそれがあります。研究ではさらに、オニヒトデと同じ生息地に生息していても同様には増殖しないアオヒトデのゲノムも解析し、このような生態の違いの根底にある遺伝学を理解することを目指しています。
また、マイヤーズ教授は、エヴァン・エコノモ教授が率いる生物多様性・複雑性研究ユニットとの共同研究も開始しました。ゲノムシーケンシングを用いてアリの進化を遡り、アリの顎、つまり下顎の形と機能がどのように進化してきたかを明らかにしようとしています。
さらに、進化ゲノミクスユニットを率いるトマ・ブーギニョン教授と共同で、シロアリの全種のゲノム解読を目標とした百科事典的な研究を進めています。
「ロングリードシーケンシングが登場したことで、今では多くの人が自然界のすべてをシーケンシングしようと意欲的になっています。そして、私もその一端を担いたいと思っています。それが全体像です」とマイヤーズ教授は述べています。

マイヤーズ教授は、OISTで勤務するのは初めてですが、幼少期の3年間を横浜で過ごしており、日本を全く知らない人ではありません。教授は、渡航制限が解除されたら実際にOISTを訪れたいと考えており、それを楽しみにしています。
「日本の人々も、文化も、食事も大好きです。そして、OISTには感銘を受けました。本当に有望な研究機関です。その一部になれることを嬉しく思っています」と語っています。
専門分野
研究ユニット
広報・取材に関するお問い合わせ
報道関係者専用問い合わせフォーム














